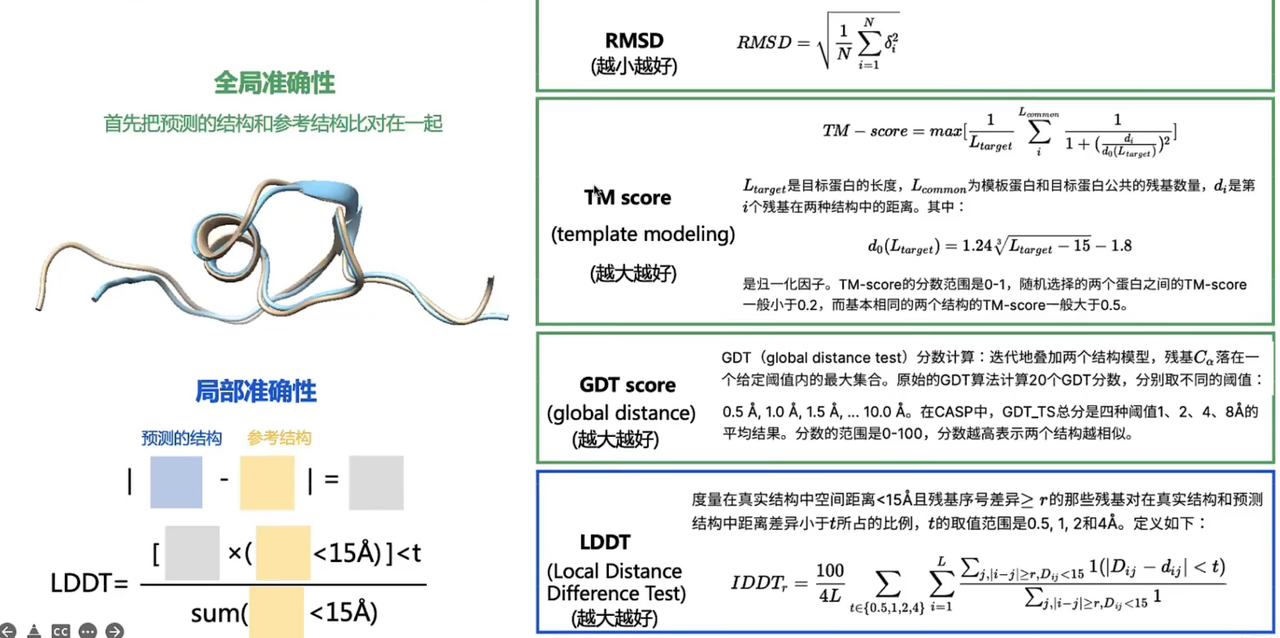
Metrics
Structure
RMSD(↓)
\[
RMSD=\sqrt{\frac{1}{N}\sum_{i=1}^N \delta_i^2}
\]
- \(N\)是原子数, \(\delta_i^2=(x_i-\hat{x_i})^2+(y_i-\hat{y_i})^2+(z_i-\hat{z_i})^2\) 表示每对ground truth原子和预测原子之间的距离。但是如何配对很关键 。 Kabsch Algorithm
\[
dRMSD=\sqrt{\frac{2}{N(N-1)}\sum_i \sum_j(d_{ij}-\hat{d_{ij}})^2}
\]
- 任意两对原子之间的距离
- 跟能量的相关性更强
TMScore(↑)
- 范围在(0,1] 1表示完全匹配。 TM-score>0.5认为结构大致相同
- 与蛋白质长度无关
GDT(↑)
LDDT(↑)
总结: RMSD,TMScore,GDT都是全局准确性